 |
Μετάφραση: Απολλόδωρος.Η επιδημία του 2019 νέου κορονοϊού ή της νόσου του κορονοϊού (COVID-19) έχει χαρακτηριστεί ως απειλή για ολόκληρο τον κόσμο από τις αρχές του 2020. Οι επιστήμονες εργάζονται νυχθημερόν για να κατανοήσουν την προέλευση της COVID-19. Ίσως έχετε ήδη ακούσει την είδηση ότι έχει δημοσιευτεί το ύποπτο γονιδίωμα του SARS-CoV-2 [1] ;...
Τώρα, με ποιον ακριβώς τρόπο οι επιστήμονες ταυτοποίησαν το πλήρες γονιδίωμα του SARS-CoV-2 ;Αυτό το άρθρο εξηγεί με καθαρά τεχνικούς όρους πώς το πέτυχαν αυτό. Αναμφίβολα θα σας γίνει σαφές ότι η μέθοδος αυτή δεν είναι κατάλληλη ούτε για τον προσδιορισμό της προέλευσης του νουκλεϊκού οξέος ενός δείγματος, ούτε για τον ακριβή προσδιορισμό ενός γονιδιώματος. Χρησιμοποιούνται διάφοροι αλγόριθμοι, οι υπολογισμοί των οποίων επιχειρούν να προτείνουν ένα πλασματικό κατασκεύασμα βάσει προδιαγραφών, το οποίο στη συνέχεια με τη σειρά του χρησιμεύει ως πρότυπο για όλες τις περαιτέρω διαδικασίες. Ωστόσο, κάθε επιμέρους αλληλούχιση οδηγεί σε διαφορετικά αποτελέσματα από την προηγούμενη αλληλούχιση, συχνά αυτές οι αποκλίσεις πλασάρονται ως «μεταλλάξεις» [2], αλλά πρόκειται απλώς για αποκλίσεις στον υπολογισμό εκ μέρους των εργαλείων ανάλυσης γονιδιώματος.
Λόγω του γεγονότος ότι
καμία δομή που ισχυρίζονται ότι είναι ιογενής δεν έχει ποτέ απομονωθεί σε καθαρή καλλιέργεια και δεν έχει πραγματοποιηθεί βιοχημικός χαρακτηρισμός απευθείας από αυτήν, αλλά κάθε αλληλούχιση βασίζεται σε ένα μείγμα γενετικού υλικού για το οποίο δεν μπορεί να προσδιοριστεί η προέλευση, όλες οι υπολογιστικά υποβοηθούμενες κατασκευές πρέπει να αντιμετωπίζονται ως καθαρή εικασία.Ως μια μικρή, αλλά όχι λιγότερο σημαντική πληροφορία εκ των προτέρων, η οποία σας καταδεικνύει άριστα ότι ο τεχνικός χειρισμός της ευθυγράμμισης/συναρμολόγησης (όπως θα εξηγηθεί στο άρθρο σε λίγο) είναι καθαρά πλασματικός και χειριστικός:
Το 70% των ιολόγων δεν διευκρινίζει ποια τεχνική συναρμολόγησης χρησιμοποιεί!
Και το ανείπωτο γεγονός είναι ότι το 100% των ιολόγων δεν αναφέρουν ποιο ποσοστό του υπολογισμένου "ιϊκού" γονιδιώματος τροποποιήθηκε μέχρι να δημοσιευθεί στη συνέχεια ως τελικό γονιδίωμα.
Τα δεδομένα του γονιδιώματος που δημοσίευσε και διέθεσε για μεταφόρτωση ο Κινέζος Fan Wu et. al [1] δεν είναι καθόλου ακατέργαστα δεδομένα, αλλά προηγουμένως παραποιημένα δεδομένα ! Το 80% των ανθρώπινων αλληλουχιών έχει εξαφανιστεί και το σημαντικότερο: ό,τι έχει δημοσιευτεί ως "ιογενές" αλλού έχει φιλτραριστεί! Αυτό καθιστά ακόμη πιο δύσκολη την ανασύνθεση της ισχυριζόμενης ανακάλυψης ενός νέου ιού που προκαλεί ασθένεια, αφού μάταια αναζητά κανείς και πειράματα ελέγχου (Control experiments) στις δημοσιευμένες εργασίες.
Αλλά τώρα ερχόμαστε στην τεχνική ανάλυση του γονιδιώματος.
ΓΟΝΙΔΙΩΜΑ
Γονιδίωμα είναι ο όρος που χρησιμοποιείται για να περιγράψει το σύνολο του γενετικού υλικού, συμπεριλαμβανομένων όλων των γονιδίων ενός οργανισμού. Το γονιδίωμα περιέχει όλες τις πληροφορίες ενός οργανισμού που είναι απαραίτητες για τη δομή και τη συντήρησή του.
· Οι ιοί που προκαλούν ασθένειες ορίζονται από το γεγονός ότι η αλληλουχία τους (γονιδίωμα) είναι μοναδική και δεν απαντάται σε υγιείς οργανισμούς.
· Προκειμένου να ανιχνευθεί και να προσδιοριστεί η παρουσία του γενετικού υλικού ενός ιού, σύμφωνα με τους νόμους της ορθής κρίσης και της λογικής που προηγούνται κάθε επιστήμης ως θεμελιώδης κανόνας, ο ιός αυτός πρέπει να απομονωθεί και να υπάρχει σε καθαρή μορφή, ώστε να μην παρερμηνεύονται οι κυτταρικές γενετικές αλληλουχίες ως συστατικά ενός ιού.
· Ο προσδιορισμός της αλληλουχίας μιας γενετικής ουσίας είναι δυνατός μόνον εάν αυτή υπάρχει με τη μορφή DNA.
ΑΛΛΗΛΟΥΧΙΣΗ
Πώς μπορούν να διαβαστούν οι πληροφορίες που υπάρχουν στο γονιδίωμα ; Σε αυτό το σημείο μπαίνει στο παιχνίδι η αλληλούχιση.
Η αλληλούχιση χρησιμοποιείται για τον προσδιορισμό της αλληλουχίας μεμονωμένων γονιδίων, ολόκληρων χρωμοσωμάτων ή ολόκληρων γονιδιωμάτων ενός οργανισμού.
 |
Ειδικά μηχανήματα που ονομάζονται μηχανές αλληλούχισης χρησιμοποιούνται για την εξαγωγή σύντομων τυχαίων αλληλουχιών από το γονιδίωμα που μας ενδιαφέρει. Οι τρέχουσες τεχνολογίες αλληλούχισης δεν μπορούν να διαβάσουν ολόκληρο το γονιδίωμα ταυτόχρονα. Διαβάζονται μικρά κομμάτια με μέσο μήκος μεταξύ 50-300 βάσεων (αλληλούχιση επόμενης γενιάς/βραχείες αναγνώσεις) ή 10.000-20.000 βάσεων (αλληλούχιση τρίτης γενιάς/μακρές αναγνώσεις), ανάλογα με την τεχνολογία που χρησιμοποιείται. Αυτά τα μικρά κομμάτια ονομάζονται reads.
· Η παρουσία και το μήκος ενός γενετικού υλικού προσδιορίζεται με το διαχωρισμό του κατά μήκος σε ηλεκτρικό πεδίο. Τα μικρά κομμάτια ταξιδεύουν γρηγορότερα, τα μεγαλύτερα κομμάτια ταξιδεύουν πιο αργά. Ταυτόχρονα, για να είναι δυνατός ο προσδιορισμός του μήκους του γενετικού υλικού που πρόκειται να εξεταστεί, προστίθενται κομμάτια γενετικού υλικού διαφορετικού μήκους και γνωστού μήκους. Αυτή η αξιόπιστη τυποποιημένη τεχνική για την ανίχνευση και τον προσδιορισμό του μήκους του γενετικού υλικού είναι γνωστή ως "ηλεκτροφόρηση πηκτής" (Gel electrophoresis).
· Εάν η συγκέντρωση ενός συγκεκριμένου DNA είναι πολύ χαμηλή ώστε να ανιχνευθεί με την τεχνική της ηλεκτροφόρησης σε πηκτή, μπορεί να ενισχυθεί με την απεριόριστη τεχνική ενίσχυσης του DNA που ονομάζεται αλυσιδωτή αντίδραση πολυμεράσης (PCR). Με τον τρόπο αυτό, μη ανιχνεύσιμο DNA μπορεί να γίνει ορατό στην ηλεκτροφόρηση σε πηκτή. Αυτό αποτελεί προϋπόθεση για να καταστεί το γενετικό υλικό προσιτό για περαιτέρω έρευνες, ιδίως για τον επακόλουθο, καθοριστικό προσδιορισμό του μήκους και της αλληλουχίας του. Η μέθοδος αυτή συντομογραφείται επίσης ως PCR.
Αν αναζητάτε πιο λεπτομερείς πληροφορίες για το πώς ακριβώς λειτουργεί η αλληλούχιση των ιικών γονιδιωμάτων από κλινικά δείγματα, θα τις βρείτε στα ακόλουθα άρθρα.
ΣΥΝΑΡΜΟΛΟΓΗΣΗ ΓΟΝΙΔΙΩΜΑΤΟΣ
Αφού είναι διαθέσιμα μικρά κομμάτια του γονιδιώματος, πρέπει να τα συνδυάσουμε με βάση τις πληροφορίες επικάλυψής τους και να συγκροτήσουμε το πλήρες γονιδίωμα. Αυτή η διαδικασία ονομάζεται συναρμολόγηση, είναι παρόμοια με την επίλυση ενός παζλ. Χρησιμοποιεί κανείς ειδικά εργαλεία λογισμικού που ονομάζονται assemblers (συναρμολογητές) για να συναρμολογήσει (να ενώσει) αυτές τις αναγνώσεις (reads) σύμφωνα με την επικάλυψή τους. Αυτό χρησιμοποιείται για να δημιουργηθούν συνεχείς συμβολοσειρές που ονομάζονται contigs. Αυτά τα contigs μπορεί να είναι ολόκληρο το ίδιο το γονιδίωμα ή μόνο κλάσματα αυτού (όπως φαίνεται στην Εικόνα 2). Το σημαντικό σημείο εδώ είναι ότι η πηγή του υλικού δεν έχει σημασία για το σκοπό αυτό.
Οι Κινέζοι ιολόγοι δεν πραγματοποίησαν πειράματα ελέγχου για να αποκλείσουν το ενδεχόμενο
· ότι ακόμη και με ανθρώπινο/μικροβιακό RNA από πλύση του πνεύμονα ενός υγιούς ατόμου,
· ενός ανθρώπου με άλλη πνευμονική νόσο
· ενός ανθρώπου που είχε αρνητική εξέταση SARS-CoV-2,
· ή από τέτοιο RNA από εφεδρικά δείγματα από την εποχή που ο ιός SARS-CoV-2 ήταν άγνωστος,
να είναι δυνατή η ίδια ακριβώς συναρμολόγηση (assembly) γονιδιώματος του ιού από μικρά τμήματα RNA!
Οι συναρμολογητές χωρίζονται σε δύο κατηγορίες ως εξής,
· De novo assemblers: συναρμολογούν χωρίς τη χρήση γονιδιωμάτων αναφοράς (π.χ.: SPAdes, SGA, MEGAHIT, Velvet, Canu και Flye).
· Συναρμολογητές καθοδηγούμενοι από αναφορές: συναρμολόγηση με αντιστοίχηση αλληλουχιών σε γονιδιώματα αναφοράς .
ΔΥΟ ΚΥΡΙΟΙ ΤΥΠΟΙ ΣΥΝΑΡΜΟΛΟΓΗΤΩΝ
Στη βιβλιογραφία της βιοπληροφορικής απαντώνται δύο κύριοι τύποι συναρμολογητών. Ο πρώτος τύπος είναι η μέθοδος συναίνεσης διάταξης επικάλυψης (OLC). Στη μέθοδο OLC, προσδιορίζονται πρώτα όλες οι επικαλύψεις μεταξύ των αναγνώσεων (reads). Στη συνέχεια, όλες οι αναγνώσεις και οι επικαλύψεις διατάσσονται με τη μορφή γραφήματος. Τέλος, προσδιορίζεται η "αλληλουχία συναίνεσης". Η SGA είναι ένα δημοφιλές εργαλείο που βασίζεται στη μέθοδο OLC.
Ο δεύτερος τύπος συναρμολογητή είναι η μέθοδος de-Bruijn graph (DBG) [3]. Αντί να χρησιμοποιεί τις πλήρεις αναγνώσεις (reads) ως έχουν, η μέθοδος DBG σπάει τις αναγνώσεις σε ακόμη μικρότερα θραύσματα που ονομάζονται k-mers (μήκους k) και στη συνέχεια δημιουργεί ένα de-Bruijn γράφημα χρησιμοποιώντας όλα τα k-mers. Τέλος, οι αλληλουχίες του γονιδιώματος συνάγονται με βάση το γράφημα de-Bruijn. Το SPAdes είναι ένας δημοφιλής συναρμολογητής που βασίζεται στη μέθοδο DBG.
ΤΙ ΜΠΟΡΕΙ ΝΑ ΠΑΕΙ ΣΤΡΑΒΑ ΣΤΗ ΣΥΝΑΡΜΟΛΟΓΗΣΗ ΓΟΝΙΔΙΩΜΑΤΟΣ;
Τα γονιδιώματα περιέχουν μοτίβα νουκλεϊκών οξέων που εμφανίζονται πολλές φορές σε όλο το γονιδίωμα. Αυτές οι δομές ονομάζονται επαναλήψεις. Αυτές οι επαναλήψεις μπορεί να περιπλέξουν τη διαδικασία συναρμολόγησης και να οδηγήσουν σε ασάφειες.
Δεν μπορούμε να εγγυηθούμε ότι ο αλληλουχιστής μπορεί να παράγει αναγνώσματα που καλύπτουν ολόκληρο το γονιδίωμα. Ο αλληλουχιστής ενδέχεται να παραλείψει ορισμένα τμήματα του γονιδιώματος και ενδέχεται να μην υπάρχουν αναγνώσεις που να καλύπτουν τη συγκεκριμένη περιοχή. Αυτό θα επηρεάσει τη διαδικασία συναρμολόγησης και αυτές οι παραλειφθείσες περιοχές δεν θα υπάρχουν στην τελική συναρμολόγηση.
Οι συναρμολογητές γονιδιώματος θα πρέπει να αντιμετωπίσουν αυτές τις προκλήσεις και να προσπαθήσουν να ελαχιστοποιήσουν τα σφάλματα που προκαλούνται κατά τη συναρμολόγηση.
ΠΩΣ ΑΞΙΟΛΟΓΟΥΝΤΑΙ ΟΙ ΣΥΝΑΡΜΟΛΟΓΗΣΕΙΣ;
Η αξιολόγηση των συναρμολογήσεων είναι πολύ σημαντική επειδή πρέπει να αποφασίσουμε αν η προκύπτουσα συναρμολόγηση πληροί τα πρότυπα. Ένα από τα πιο δημοφιλή και ευρέως χρησιμοποιούμενα εργαλεία αξιολόγησης συναρμολόγησης είναι το QUAST. Παρακάτω παρατίθενται ορισμένα κριτήρια που χρησιμοποιούνται για την αξιολόγηση των συναρμολογήσεων.
· N50: ελάχιστο μήκος contig που απαιτείται για να καλύψει το 50% του συνολικού μήκους του assembly.
· L50: αριθμός contigs μεγαλύτερου μήκους από το N50.
· NG50: ελάχιστο μήκος contig που απαιτείται για την κάλυψη του 50% του μήκους του γονιδιώματος αναφοράς.
· LG50: αριθμός contigs μεγαλύτερου μήκους από το NG50
· NA50: ελάχιστο μήκος ευθυγραμμισμένων μπλοκ που απαιτείται για την κάλυψη του 50% του συνολικού μήκους της συνέλευσης
· LA50: αριθμός contigs μεγαλύτερου μήκους από NA50
· Ποσοστό γονιδιώματος (%): ποσοστό βάσεων που ταιριάζουν με το γονιδίωμα αναφοράς.
ΕΚΤΕΛΩΝΤΑΣ ΤΗΝ «ΒΡΩΜΙΚΗ» ΔΟΥΛΕΙΑ
Ας ξεκινήσουμε με τα πειράματα. Θα χρησιμοποιήσω τον συναρμολογητή SPAdes για να συναρμολογήσω τις αναγνώσεις που προέρχονται από αλληλουχισμένα δείγματα ασθενών. Ο SPAdes χρησιμοποιεί αναγνώσματα αλληλούχισης επόμενης γενιάς. Μπορείτε επίσης να μεταφορτώσετε («κατεβάσετε») δωρεάν το QUAST. Μπορείτε να λάβετε τον κώδικα και τα δυαδικά αρχεία από τις αντίστοιχες ιστοσελίδες και να «τρέξετε» αυτά τα εργαλεία.
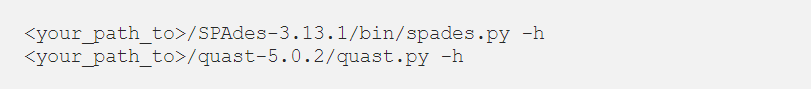
Εισάγετε τις ακόλουθες εντολές και επαληθεύστε ότι τα εργαλεία λειτουργούν σωστά.
«ΚΑΤΕΒΑΣΤΕ» ΤΑ ΔΕΔΟΜΕΝΑ
Υποθέτω ότι γνωρίζετε πώς να κατεβάζετε δεδομένα από το Εθνικό Κέντρο Βιοτεχνολογικών Πληροφοριών (NBCI). Εάν όχι, μπορείτε να ανατρέξετε σε αυτόν τον σύνδεσμο.
Οι αναγνώσεις για τα πειράματά μας μπορούν να μεταφορτωθούν από το NCBI χρησιμοποιώντας τον αριθμό πρόσβασης NCBI SRX7636886. Μπορείτε να κατεβάσετε την εκτέλεση SRR10971381, η οποία περιέχει αναγνώσματα που προέρχονται από μια εκτέλεση Illumina MiniSeq. Φροντίστε να κατεβάσετε τα δεδομένα σε μορφή FASTQ. Μπορείτε να βρείτε το αρχείο που κατεβάσατε ως "sra_data.fastq.gz". Μπορείτε να εξαγάγετε το αρχείο FASTQ χρησιμοποιώντας το gunzip.
Μετά την εξαγωγή, μπορείτε να εκτελέσετε την ακόλουθη εντολή bash για να μετρήσετε τον αριθμό των αναγνώσεων στο σύνολο δεδομένων μας. Θα δείτε ότι υπάρχουν 56.565.928 αναγνώσεις.
Μπορείτε να κατεβάσετε το δημοσίως διαθέσιμο πλήρες γονιδίωμα SARS Cov-2 [1] από το NCBI με αριθμό πρόσβασης GenBank MN908947 (να θυμάστε ότι δεν πρόκειται για τα πραγματικά ακατέργαστα δεδομένα). Θα δείτε ένα αρχείο σε μορφή FASTA. Αυτό θα είναι το γονιδίωμα αναφοράς μας. Σημειώστε ότι έχει μετονομαστεί σε "MN908947.fasta".
Συναρμολόγηση
Ας συναρμολογήσουμε τις αναγνώσεις από το COVID-19. Εκτελέστε την ακόλουθη εντολή για να συναρμολογήσετε τις αναγνώσεις με το SPAdes. Μπορείτε να περάσετε το συμπιεσμένο αρχείο .gz απευθείας στο SPAdes.
Εδώ έχουμε χρησιμοποιήσει τον γενικό συναρμολογητή SPAdes ως επίδειξη για αυτό το άρθρο. Ωστόσο, δεδομένου ότι το σύνολο δεδομένων reads αποτελείται από δεδομένα RNA-Seq, είναι προτιμότερο να χρησιμοποιήσετε την επιλογή "--rna" στο SPAdes.
Στο φάκελο εξόδου, θα δείτε ένα αρχείο με όνομα "contigs.fasta" που περιέχει τα τελικά συναρμολογημένα contigs μας.
ΑΞΙΟΛΟΓΗΣΗ ΤΩΝ ΑΠΟΤΕΛΕΣΜΑΤΩΝ ΤΗΣ ΣΥΝΑΡΜΟΛΟΓΗΣΗΣ
Εκτελέστε το QUAST στις συναρμολογήσεις χρησιμοποιώντας την ακόλουθη εντολή.
 |
ΕΜΦΑΝΙΣΗ ΤΟΥ ΑΠΟΤΕΛΕΣΜΑΤΟΣ ΤΗΣ ΑΞΙΟΛΟΓΗΣΗΣ
Όταν το QUAST ολοκληρωθεί, μπορείτε να μεταβείτε στο φάκελο «quastResult» και να δείτε τα αποτελέσματα της αξιολόγησης. Μπορείτε να προβάλετε την έκθεση QUAST ανοίγοντας το αρχείο report.html στο πρόγραμμα περιήγησης στο διαδίκτυο. Θα δείτε μια αναφορά παρόμοια με αυτή που φαίνεται στην Εικόνα 3. Μπορείτε να κάνετε κλικ στην επιλογή "Advanced Report" (Έκθεση για προχωρημένους) για να λάβετε περισσότερες πληροφορίες, όπως NG50 και LG50.
Μπορείτε να εξετάσετε τις τιμές των διαφόρων κριτηρίων βαθμολόγησης, όπως η αναλογία γονιδιώματος NG50, NA50, οι λανθασμένες συναρμολογήσεις και ο αριθμός των contigs. Επίσης, μπορείτε να προβάλετε τη στοίχιση του contig στο γονιδίωμα αναφοράς χρησιμοποιώντας το πρόγραμμα περιήγησης contig του Icarus (κάντε κλικ στο "View in Icarus contig browser"), όπως φαίνεται στην Εικόνα 4.
Στο πρόγραμμα περιήγησης contig του Icarus, μπορούμε να δούμε ότι το contig με την ονομασία "NODE_1" ταιριάζει πολύ στενά με το γονιδίωμα αναφοράς του COVID-19. Έχει περιεκτικότητα γονιδιώματος 99,99% (όπως φαίνεται στην Εικόνα 3). Επιπλέον, το συνολικό ευθυγραμμισμένο μήκος των 29.900 ζευγών βάσεων είναι πολύ κοντά στο μήκος του γονιδιώματος αναφοράς, το οποίο είναι 29.903 ζεύγη βάσεων.
ΟΠΤΙΚΟΠΟΙΗΣΗ ΤΟΥ ΔΙΑΓΡΑΜΜΑΤΟΣ ΣΥΝΑΡΜΟΛΟΓΗΣΗΣ
Υπάρχει ένα εργαλείο που ονομάζεται Bandage και σας επιτρέπει την οπτικοποίηση του διαγράμματος συναρμολόγησης. Μπορείτε να κατεβάσετε τα προμεταγλωττισμένα δυαδικά αρχεία από την αρχική τους σελίδα και να εκτελέσετε το εργαλείο. Μπορείτε να φορτώσετε το αρχείο γραφήματος "assembly_graph_with_scaffolds.gfa" που βρίσκεται στο φάκελο εξόδου του SPAdes (Μεταβείτε στο File → Load Graph → επιλέξτε το αρχείο .gfa στο Output και ανοίξτε το) στο Bandage και κάντε κλικ στο "Draw graph" για να το οπτικοποιήσετε όπως φαίνεται στην Εικόνα 5. Σημειώστε ότι το μακρύ, πράσινου χρώματος, καμπυλωτό τμήμα στη μέση της πρώτης σειράς τμημάτων στο Σχήμα 5 αντιστοιχεί στο NODE_1 της συναρμολόγησής μας SPAdes.
 |
Πρακτικό παράδειγμα από το towardsdatascience
ΠΩΣ ΥΠΟΛΟΓΙΣΤΗΚΕ ΓΙΑ ΠΡΩΤΗ ΦΟΡΑ ΤΟ ΓΟΝΙΔΙΩΜΑ ΤΟΥ SARS-COV-2 ;
Τώρα που το γονιδίωμα αναφοράς του SARS-CoV-2 είναι διαθέσιμο, μπορούμε να αξιολογήσουμε τη συναρμολόγησή μας. Ωστόσο, αρχικά δεν υπήρχε ακριβές γονιδίωμα αναφοράς για τον SARS-CoV-2, οπότε τι έκαναν οι επιστήμονες για να το βρουν ;
Όπως εξηγείται σε αυτό το άρθρο, η ανάλυση του ιϊκού γονιδιώματος εμπίπτει στη μεταγονιδιωματική και υπάρχουν πολλές τεχνικές για να γίνει αυτό. Ανέλυσαν την κάλυψη των contigs (ο μέσος αριθμός των αναγνώσεων που καλύπτουν κάθε θέση βάσης σε ένα contig) και το συνέκριναν με την απομόνωση*** (isolate) του SARS-like coronavirus (CoV) της νυχτερίδας - νυχτερίδα SL-CoVZC45 (GenBank accession number MG772933) [3] [6]. Τα αποτελέσματα έδειξαν ότι το μακρύτερο συναρμολογημένο contig τους είχε "υψηλή" κάλυψη (από τη δική μας συναρμολόγηση μπορείτε να δείτε ότι το NODE_1 έχει επίσης υψηλή τιμή κάλυψης) και ήταν πολύ στενά συνδεδεμένο με το SL-CoVZC45 της νυχτερίδας.
[*** isolate: απομόνωση ονομάζεται ένας ιός που έχει «απομονωθεί» από έναν μολυσμένο ξενιστή και πολλαπλασιασθεί σε καλλιέργεια ]
Οι Κινέζοι ιολόγοι επισημαίνουν μάλιστα ρητά ότι η κατασκευασμένη αλυσίδα γονιδιώματος έχει έως και 90% ομοιότητα με αλυσίδες γονιδιώματος ακίνδυνων και αβλαβών κορονοϊών στις νυχτερίδες που είναι γνωστές εδώ και δεκαετίες.
Αυτή η ομοιότητα 90% προκύπτει από το γεγονός ότι ακριβώς ένα τέτοιο "γονιδίωμα" ενός ακίνδυνου "ιού κορώνας" νυχτερίδας χρησιμοποιήθηκε ως πρότυπο για την ευθυγράμμιση των πολυάριθμων πολύ σύντομων αλληλουχιών γονιδίων (= ευθυγράμμιση). Όπου πρέπει να ειπωθεί ότι αυτό το γονιδίωμα της νυχτερίδας, όπως και όλα τα "γονιδιώματα" των "ιών" που φέρονται να προκαλούν ασθένειες, μόνον υπολογίστηκε, δηλαδή νοητά συναρμολογήθηκε από πολύ σύντομες ενδογενείς αλληλουχίες γονιδίων ή/και από γονιδιακά θραύσματα πολυάριθμων μικροβίων, τα οποία στην πραγματικότητα δεν βρέθηκαν ποτέ ως ολόκληρος κληρονομικός κλώνος και επίσης δεν εμφανίζονται ως πλήρες " ιϊκό γονιδίωμα" πουθενά στην επιστημονική βιβλιογραφία.
Έτσι, η μελέτη της 24.01.2020 αναφέρει στην ενότητα "Συζήτηση" (Discussion):
"Η μελέτη μας δεν πληροί τα αξιώματα του Koch" (Our study does not fulfill Koch’s postulates).
Είναι σημαντικό να σημειωθεί ότι δεν είναι γνωστό ποιο ποσοστό του υπολογισμένου "ιικού" γονιδιώματος τροποποιήθηκε πριν δημοσιευθεί ως τελικό γονιδίωμα.
Η δημοσίευση των Fan Wu et al, στο Nature, Vol. 579 της 3ης Φεβρουαρίου 2020, στην οποία παρουσιάστηκε για πρώτη φορά το γονιδίωμα (πλήρης αλυσίδα γονιδιώματος) του SARS-CoV-2 και προωθήθηκε ώστε να γίνει το πρότυπο για όλες τις περαιτέρω ευθυγραμμίσεις, έδειξε ότι χρησιμοποιήθηκε σαφώς ολόκληρο το RNA που ελήφθη από το βρογχικό έκπλυμα (BALF) ενός ασθενούς, χωρίς να προηγηθεί οποιαδήποτε απομόνωση ή εμπλουτισμός των ιϊκών δομών ή νουκλεϊκών οξέων.
Σε αυτή τη δημοσίευση, ο καθηγητής Zhang περιγράφει πώς υπολόγισε ένα γονιδίωμα 29.903 νουκλεοτιδίων χρησιμοποιώντας σύντομα γονιδιακά τμήματα μήκους μόνον 21 και 25 νουκλεοτιδίων (αυτές είναι οι προεπιλεγμένες παράμετροι στα προγράμματα ευθυγράμμισης Megahit και Trinity που χρησιμοποίησε) με βάση μια δεδομένη αλληλουχία ενός γονιδιώματος (αβλαβής ιός της κορώνας της νυχτερίδας) χρησιμοποιώντας επτά διαφορετικές, πολύ περίπλοκες μεθόδους, συμπεριλαμβανομένων στατιστικών μεθόδων.
Το RNA αυτό μετατράπηκε στη συνέχεια σε cDNA και μόρια μήκους μόλις 150 νουκλεοτιδίων αλληλουχήθηκαν προκειμένου να κατασκευαστεί το πλήρες γονιδίωμα μήκους περίπου 30.000 νουκλεοτιδίων αποκλειστικά με υπολογισμό (in silico).
Υποθέτει - χωρίς να το δηλώνει ρητά - ότι οι σύντομες αλληλουχίες από τις οποίες προσθέτει την προτεινόμενη αλληλουχία του γονιδιώματος του ιού SARS-CoV-2 είναι ιϊκής φύσης, διότι αποκλείει τις μακρύτερες αλληλουχίες που προκύπτουν από την επικάλυψη (= contigs) των σύντομων κομματιών 21 και 25 και οι οποίες έχουν ομοιότητα με τις ανθρώπινες αλληλουχίες από τη μεταγενέστερη προσθήκη στο ιϊκό γονιδίωμα.
Με απλά λόγια, αυτό σημαίνει ότι:
Εφόσον οι "γνωστές" σε εμάς ανθρώπινες αλληλουχίες έχουν αφαιρεθεί/απομακρυνθεί από το μείγμα του γενετικού υλικού, οι εναπομείνασες αλληλουχίες, γέννημα ψυχαναγκαστικής καλωδιακής/ηλεκτρονικής σκέψης (η οποία επισφραγίστηκε το 1954 με την απονομή του βραβείου Νόμπελ στον John Franklin Enders για μια εικασία) [7], θεωρούνται ότι πρέπει να είναι ιϊκής φύσης.
Ποιο ποσοστό ολόκληρου του γονιδιώματος έχει κενά (= 1 % έως σχεδόν τα πάντα;;;) δεν αναφέρεται.
ΛΟΓΙΚΗ ΣΥΝΕΠΕΙΑ:
Αυτό που έχει δημιουργηθεί τεχνητά εδώ σε όλα τα είδη των βημάτων, όλα κάτω από απλώς θεωρούμενες, ποτέ επαληθευμένες "υποθέσεις", δεν έχει απολύτως ΚΑΜΙΑ σχέση με την πραγματικότητα!
Η μέθοδος αλληλούχισης δεν μπορεί ούτε να πει από ποια υποτιθέμενη πηγή προέρχεται το γονιδίωμα που δημιουργήθηκε με πολλά βήματα υπολογισμού, ούτε ότι πρόκειται καθόλου για αμιγώς ιογενή φύση.
Είναι το πολύ-πολύ ένα εργαλείο δημιουργίας ενός νέου πλασματικά κατασκευασμένου σκέλους γονιδιώματος από πολλές πολύ μικρές αλληλουχίες γονιδίων, μέσω διαφόρων αλγορίθμων και των λεγόμενων προγραμμάτων συμπλήρωσης κενών (κλείνει τα κενά στην κατασκευή του γονιδιώματος).
Είναι κάτι παραπάνω από θρασύ να υποστηρίζεται ότι ακριβώς αυτή η κατασκευή (του γονιδιώματος), η οποία έγινε από ένα δείγμα μέσω του BALF ενός ασθενούς χωρίς απομόνωση μιας συγκεκριμένης δομής, είναι ιογενής μόνο και μόνο επειδή ο ασθενής παρουσίασε συμπτώματα.
Οι βιοπληροφορικοί δεν ενδιαφέρονται για την πηγή της γονιδιακής αλληλουχίας
Όταν πρωτοσυνελήφθη η ιδέα των γενετικών αλυσίδων των ιών της γρίπης, έμβρυα κοτόπουλου τραυματίστηκαν μηχανικά και δηλητηριάστηκαν, και ένα μοντέλο δημιουργήθηκε επίπονα με το χέρι από τις σύντομες αλληλουχίες νουκλεϊκών οξέων του ιστού που πέθαινε. Σήμερα, αυτό γίνεται από προγράμματα ηλεκτρονικών υπολογιστών που τροφοδοτούνται με τις αλληλουχίες που οι ιολόγοι ισχυρίζονται ότι είναι "ιογενείς".
Το από πού προέρχονται στην πραγματικότητα αυτές οι αλληλουχίες είναι αδιάφορο για τους βιοπληροφορικούς που δημιουργούν τα γονιδιώματα των πλασματικών ιών με "ευθυγράμμιση".
Παρεμπιπτόντως, ο Erwin Chargaff προειδοποίησε για αυτή την εξέλιξη ήδη από το 1976 στο βιβλίο του: "Η φωτιά του Ηράκλειτου" (Heraclitean Fire: Sketches from a Life before Nature).
Πηγές:
[1] F. Wu, S. Zhao, B. Yu et al. A new coronavirus associated with human respiratory disease in China. Nature (2020). https://doi.org/10.1038/s41586-020-2008-3
[2] Die behauptete SARS-CoV-2-Mutation aus England ist eine Mogelpackung
[3] Zhenyu Li et al. Comparison of the two major classes of assembly algorithms: overlap–layout–consensus and de-bruijn-graph, Briefings in Functional Genomics, Volume 11, Issue 1, January 2012, Pages 25–37. https://doi.org/10.1093/bfgp/elr035
[4] Jang-il Sohn and Jin-Wu Nam. The present and future of de novo whole-genome assembly. Briefings in Bioinformatics, Volume 19, Issue 1, January 2018, Pages 23–40. https://doi.org/10.1093/bib/bbw096
[5] S. Heerema and C. Dekker. Graphene nanodevices for DNA sequencing. Nature Nanotech 11, 127–136 (2016). https://doi.org/10.1038/nnano.2015.307
[6]Fan Wu: A new coronavirus associated with human respiratory disease in China
Η οργάνωση του ιικού γονιδιώματος του WHCV προσδιορίστηκε με ευθυγράμμιση αλληλουχιών με δύο αντιπροσωπευτικά μέλη του γένους Betacoronavirus: έναν κοροναϊό που σχετίζεται με τον άνθρωπο (SARS-CoV Tor2, αριθμός καταχώρησης στη GenBank AY274119) και έναν κοροναϊό που σχετίζεται με νυχτερίδες (νυχτερίδα SL-CoVZC45, αριθμός καταχώρησης στη GenBank MG772933).
[7] Machtwerk - Einstieg in die Widerlegung der Virusbehauptung
Δικτυογραφία:
Fehldeutung Virus – wie durch die Genomanalyse fiktive Viren erschaffen werden – Telegraph








Δεν υπάρχουν σχόλια:
Δημοσίευση σχολίου